COVID-19 EMERGENCY
Webmaster
COVID-19 PROTOKOLOA
SARS-COV-2 BIRUSA RNA HAUSPEATUZ ETA RT-Q-PCR TEKNIKA ERABILIZ DETEKTATZEKO PROTOKOLOA
Versión SOP 1.3, 19 Abril 2020
I. Lagina hartzea
- Lagina hartzeko, bi bastoitxo erabiltzen dira, frotis nasofaringeo eta frotis faringeo bana egiteko.
- Bi bastoitxoak hodi biltzailean sartzen dira. Hodiak hazkuntza-inguruaren 2 ml izango ditu (DMEM, UTM edo baliokidea).
- Hodia itxi, eta kanpotik garbitzen da % 10eko lixiba-disoluzio batez.
II. Lagina inaktibatzea
(15-30 min, gutxi gorabehera)
- Lagina inaktibatzeko, presio negatiboko laborategi bateko BS2 ganberan edo BS3 ganberan sartzen da.
- Laginak dituen hodia ganberan sartu aurretik, berriro garbitzen da kanpotik, % 10eko lixiba-disoluzioaz.
- Bastoitxoak hazkuntza-inguruan astintzen dira, lagina ingurura pasatzen dela ziurtatzeko.
- Ganbera barruan gaudelarik, 325 μl lagin hartzen dira, eta 1,5 ml-ko hodi batera pasatzen dira. Hodiak lisirako disoluzio indargetzaile baten bolumen bera izan behar du (COBAS omni lysis buffer, Ref. 06997538190 edo Viral Lysis Buffer, NZYTech, Ref. MB40801). Guztira, 650 μl izan behar ditu hodiak.
- Azkenik eta hodiak ganberatik atera aurretik, berriro garbitzen dira % 10eko lixiba-disoluzioaz.
III. RNA hauspeatzea
(Ordubete, gutxi gorabehera)
- S2 ganbera 20 minutuz esterilizatzen da UV argia erabiliz.
- 12.000 g-an zentrifugatzen da, 10 minutuz, 4°C-an.
- Gainjalkinaren 600 μl hartu, eta 1,5 ml-ko hodi batera pasatzen da. Hodiak 600 μl isopropanol eta 4 μl glycoblue hotza (–20°C) izan behar ditu (Stocka: 15 mg/ml; Erabilera: 50 μg/ml; Invitrogen #AM9515).
- Alderantziz jarriz nahasi, eta 10 minutuz inkubatzen da izotzetan.
- 12.000 g-an zentrifugatzen da, 10 minutuz, 4°C-an.
- Gainjalkina kendu.
- % 75eko etanol hotzetik (–20 °C) 500 μl gehitzen dira.
- 12.000 g-an zentrifugatzen da, 5 minutuz, 4°C-an.
- Etanola kendu, eta pelleta airean lehortzen uzten da 5-10 minutuz.
- Pelleta berriro esekitzen da 40 μl H2O-tan (erabili biologia molekularrerako kalitatea duen ura, DNAsarik eta RNAsarik gabea eta DEPCarekin tratatu gabea), eta 60°C-an berotzen da 5 minutuz.
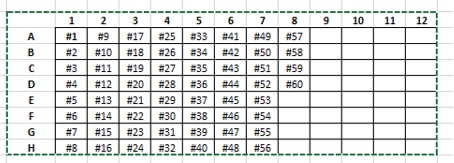
- 96 gelaxkako plaka batera pasatzen da (plakaren nomenklatura: “data-operadoreareninizialak-plaka-zk.”: 090420-DM-P1), eta itsasgarri batez estaltzen da plaka. Berehala erabiltzekoa ez bada, hotzean gordetzen da (–80°C). Berehala erabiltzekoa bada Q-PCRa zuzenean muntatzeko, izotzetan edukitzen da.
IV. RT-Q-PCR teknika egitea, TaqMan eta 384 gelaxkako plakak erabiliz
(3 ordu, gutxi gorabehera)
- Material hauek erabiltzen dira: Q-PCR one-step-erako Master nahastea [NZYTech Speedy One-step RT-qPCR Probe Master Mix (2x, NZYTech #MB40503)] eta TaqMan CDC 2019-nCoV RUO Kit zundak (hauek bakarrik: N1, N2 eta RNAseP, CDC #225397445).
- Erreakzioaren bukaerako bolumena 10 μl izaten da (8 μl TaqMan nahaste + 2 μl RNA lagin), 384 gelaxkako plaken kasuan. TaqMan nahastea honela egiten da (bolumenak gelaxkako dira):
- El volumen final de la reacción será de 10 µl (8 µl mix TaqMan + 2 µl muestra RNA) para placas de 384 pocillos. El mix TaqMan se realizará de la siguiente manera (volúmenes por pocillo):
- 5 μl NZY master nahaste (2x)
- 0,75 μl zunda
- 2,25 μl H2O (DNAsarik eta RNAsarik gabea, DEPCarekin tratatu gabea)
-
Diagnostikatu nahi diren lagin bakoitzeko bi test egingo dira, eta, horiez gain, beste kontrol hauek ere eransten dira:
- NTC: Non template control - TaqMan nahastea + 2 μl H2O (DNAsarik eta RNAsarik gabea, DEPCarekin tratatu gabea)
- Kontrol negatiboa: (HeLa-RNA edo infektatu gabeko edozein giza zelularen RNA-kultura).
- Kontrol positiboa: TaqMan nahastea + 2 μl 2019-CoV CDCren kontrol-plasmidoa (200.000 kopia/μl; CDC #225397446).
Diluzioak: 10^-2, 10^-4, 10^-5 (4.000 – 40 – 4 kopia gelaxkako, hurrenez hurren), zundek gene bakoitzeko zer sentikortasun-muga duten ikusteko. Diluzioak egiteko, DNAsarik eta RNAsarik gabeko eta DEPCarekin tratatu gabeko H2O-a erabiltzen da.
- Plaka diseinatzea eta prestatzea:
- BS2 ganbera 20 minutuz esterilizatzen da UV argia erabiliz.
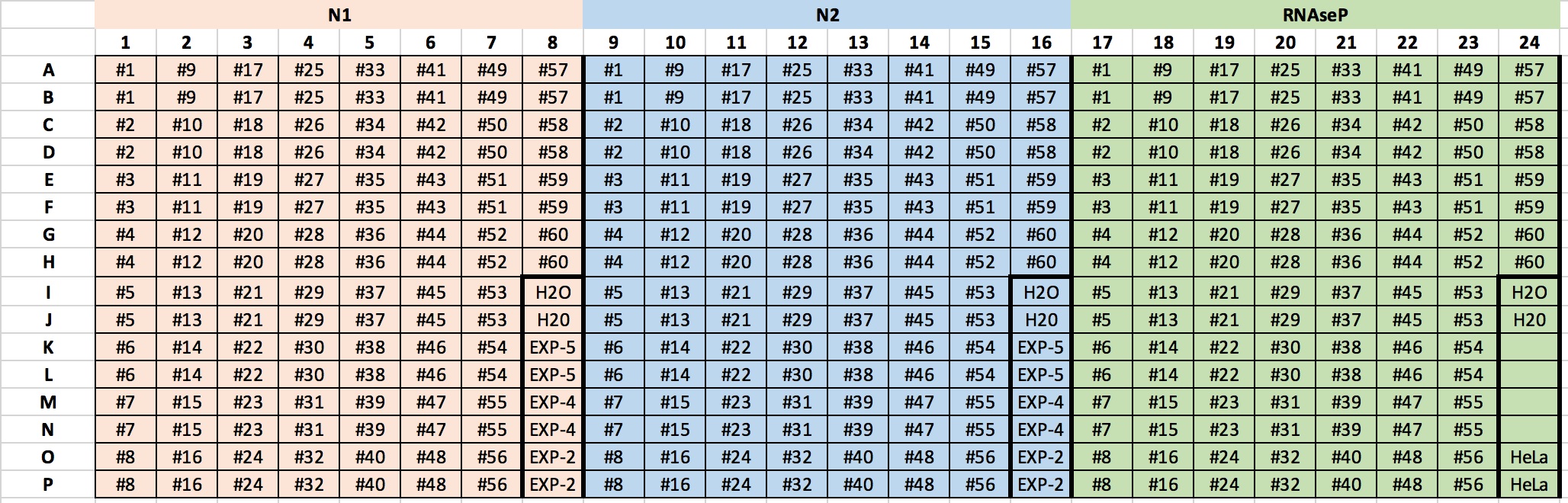
- 60 lagin bikoiztu gehi kontrolak dituen plaka bat prestatzeko, hiru nahaste prestatzen dira, zunda bakoitzeko bat (N1, N2 eta RNaseP), honako bolumen hauek erabilita:
- 800 µl TaqMan nahaste
- 120 µl zunda
-
360 µl H2O
- Si se va a pipetear con la multicanal, se pasan las mezclas a tiras de tubos de 8 (160 µl por tubo).
- Multikanalarekin pipeteatu behar bada, 8 hodiko zerrendetara pasatzen dira nahasteak (160 μl hodiko)
- Gelaxka bakoitzean, nahastearen 8 μl jartzen dira.
- Ondoren, laginaren edo kontrolaren 2 μl pipeteatzen dira bi aldiz, dagozkien gelaxketan. Estalki itsasgarri batekin estaltzen da plaka (Q-PCR sistemarekin bateragarria den optika), eta berriro zentrifugatzen da.
- Ondoren, anplifikazio-programa hau erabiltzen da BioRad CFX384touch termozikladore batean, threshold automatikoaren modua* erabiliz:
| Zikloak | Tenperatura | Denbora | Erreakzioa |
|---|---|---|---|
| 1 | 50°C | 20 min | Erretrotranskripzioa |
| 1 | 95°C |
3 min |
Polimerasaren aktibazioa |
| 40 | 95°C | 5 s | Desnaturalizazioa |
| 55°C | 50 s | Hibridazioa + luzapena |
* threshold eta baseline automatikoaren modua erabili arren, kurbak aztertu egin behar dira emaitzak esportatu aurretik, gerta baitaiteke emaitzak distortsionatzen dituen background asko egotea. Modua alda daiteke, behar izanez gero.
- Emaitzen esportazioa:
- Plaka osoaren datuak bidali behar dira beti, plakak gelaxka hutsak baditu ere
- Analisi-taldeari fitxategi hauek bidaltzen zaizkio posta elektronikoz:
- Softwarearena (*.pcrd)
- Ct balioak dituen taula (Excel y csv)
V. Emaitzak analizatzea (irizpideak)
Protokolo honetan, COVID-19aren diagnostikorako esperimentuko kontrol eta lagin klinikoen emaitzak interpretatzeko irizpideak laburbiltzen dira. 1. fasean hartu zen irizpide hori, eta CDCk argitaratutako protokoloari jarraituz eraldatu da.
Laginak: 60 lagin erauzi dira, isopropanol bidez hauspeatzeko protokoloa erabilita.
Zundak: N1, N2 eta RNAseP
Kontrolak:
- NTC: H2O (DNAsarik eta RNAsarik gabea, DEPCarekin tratatu gabea)
- Kontrol negatiboa: HeLa-RNA (edo infektatu gabeko edozein giza zelularen RNA-kultura)
- Kontrol positiboa: 2019-CoV CDCren kontrol-plasmidoa (200.000 kopia/μl).
Diluzioak: 10^-2 (CDCk gomendatzen duen 1.000ren ingurukoa), 10^-4, 10^-5 (gure detekzio-mugatik gertukoa). Diluzioak egiteko, DNAsarik eta RNAsarik gabeko eta DEPCarekin tratatu gabeko H2O-a erabiltzen da.
Control de Calidad:
- NTC (H2O) kontrolak 3 saiakuntzetan izan behar du negatibo
- Lagin kliniko eta kontrol negatibo guztietan, RNAseP anplifikatzearen emaitzak Ct < 35 izan behar du
- Kontrol positiboak N1 eta N2 saiakuntzetan anplifikatu behar du, diluzio guztietan (10^-2, 10^-4, 10^-5).
EMAITZEN INTERPRETAZIOA:
- Laginaren osotasuna baliozkotzeko, RNPase genea erabiltzen da kontrol gisa.
- RNPase-ren emaitza 35 ziklo baino gehiago (edo zehaztugabea) bada, errepikatu egin behar da SYBR erabilita.
- RNPase-ren kasuan positibo diren laginak (35 ziklo baino gutxiago) positibotzat jotzen dira baldin eta 4 kopia baino gehiago badituzte N1 zundan, N2 zundan edo bietan.
- N1 eta N2 zundetan 40 ziklo baino gehiago (eta RNPase-ren kasuan 35 ziklo baino gutxiago) ematen duten laginak negatibotzat jotzen dira.
- N1 eta N2 zundetan 4 kopia baino gutxiago (baina 40 ziklo baino gutxiago) ematen duten laginak zalantzazkoak izaten dira. Zalantzazkoak dira, orobat, erreplika tekniko inkongruenteak ematen dituzten laginak (erreplika batean, 4 kopia baino gehiago, eta bestea negatiboa (Ct>40)). Halakoetan, anplifikazio-kurba aztertu ondoren ezin bada egiaztatu artefaktu teknikorik izan ez denik, laginak SYBR metodoarekin prozesatu behar dira (VI. atalean deskribatua).
VI. Zalantzazko laginak baliozkotzea RT-Q-PCR teknika eta SYBR konposatua erabiliz
(3 ordu, gutxi gorabehera)
Zalantzazko kasuak SYBR Green eta Won et al 2020 artikuluko abiarazle multzoa erabiliz errepikatzen dira.
- Material hauek erabiltzen dira: NZYSpeedy One-step-erako Master nahastea [RT-qPCR Green Kit (NZYTech #MB34602)] eta Won et al 2020 artikuluko abiarazleak (N1, S1, RdRP eta RPP30)::
| Espeziea | Itu-genea | Abiarazlea | Forward abiarazlea (5’-3’) | Reverse abiarazlea (5’-3’) |
|---|---|---|---|---|
| SARS-CoV-2 | RdRP | SARS-CoV- 2_IBS_RdRP1 | CATGTGTGGCGGTTCACTAT | TGCATTAACATTGGCCGTGA |
| S | SARS-CoV- 2_IBS_S1 | CTACATGCACCAGCAACTGT | CACCTGTGCCTGTTAAACCA | |
| N | SARS-CoV-2_IBS_N1 | CAATGCTGCAATCGTGCTAC | GTTGCGACTACGTGATGAGG | |
| Gizakia | RPP30 | IBS_RPP30 | CTATTAATGTGGCGATTGACCGA | TGAGGGCACTGGAAATTGTAT |
- Erreakzioaren bukaerako bolumena 10 μl izaten da (8 μl SYBR Green nahaste + 2 μl RNA lagin), 384 gelaxkako plaken kasuan. Nahastea honela egiten da (bolumenak gelaxkako dira):
- 5 μL SYBR Green Master nahaste (2x)
- 1 μl abiarazle zuzenaren eta alderantzizkoaren nahastetik (5mM)
- 0.4 μl NZYRT nahaste
- 1,6 μl H2O (DNAsarik eta RNAsarik gabea, DEPCarekin tratatu gabea)
- Se añadirán 8 μl de la mezcla por pocillo
- Gelaxka bakoitzean, nahastearen 8 μl jartzen dira.
- Ondoren, laginaren edo kontrolaren 2 μl pipeteatzen dira bi aldiz, dagozkien gelaxketan. Estalki itsasgarri batekin estaltzen da plaka (Q-PCR sistemarekin bateragarria den optika), eta berriro zentrifugatzen da.
- Oharra: 96 gelaxkako plaka erabiliz gero, bolumen hau behar da erreakziorako (bolumenak gelaxkako dira):
- Posteriormente, se pipetearán 2 μl de la muestra o control por duplicado en los pocillos correspondientes, se tapará la placa con una tapa adhesiva (óptica compatible con el sistema de Q-PCR) y se centrifugará de nuevo.
- 7,5 μL SYBR Green Master nahaste (2x)
- 1,5 μl, abiarazle zuzenaren eta alderantzizkoaren nahastetik (5mM)
- 0,6 μl NZYRT nahaste
- 3,4 μl H2O (DNAsarik eta RNAsarik gabea, DEPCarekin tratatu gabea)
- Ondoren, Life Technologies QS5 (Applied) anplifikazio-programa hau erabiltzen da, threshold automatikoaren modua erabiliz::
| Zikloak | Tenperatura | Denbora | Erreakzioa |
|---|---|---|---|
| 1 | 50°C | 20 min | Erretrotranskripzioa |
| 1 | 95°C | 3 min | Polimerasaren aktibazioa |
| 40 | 95°C | 5 s | Desnaturalizazioa |
| 60°C | 50 s | Hibridazioa + luzapena |
- Diagnostikatu nahi diren laginak gehitzeaz gainera (bina testeatzeko beste), beste kontrol hauek ere eransten dira:
- NTC: Non template control - SYBR nahastea + 2 μl H2O (DNAsarik eta RNAsarik gabea, DEPCarekin tratatu gabea)
- Kontrol negatiboa: (HeLa-RNA edo edozein giza zelularen kultura ez infektatuaren RNA)
- Kontrol positiboa: 2019-CoV CDCren kontrol-plasmidoa (200.000 kopia/μl).
Diluzioak: 10^-2, 10^-3, 10^-4, 10^-5 (4.000 – 400 – 40 – 4 kopia), abiarazleek gene bakoitzeko zer sentikortasun-muga duten ikusteko. Diluzioak egiteko, DNAsarik eta RNAsarik gabeko eta DEPCarekin tratatu gabeko H2O-a erabiltzen da..
- Emaitzen esportazioa:
- Plaka osoaren datuak bidali behar dira beti, plakak gelaxka hutsak baditu ere.
- Analisi-taldeari fitxategi hauek bidaltzen zaizkio posta elektronikoz:
- Softwarearena (*.pcrd)
- Ct balioak dituen taula (Excel y csv)
EMAITZEN INTERPRETAZIOA:
- N, S eta RdpP birus-geneak aztertzen dira.
- Laginaren osotasuna baliozkotzeko, RPP30 giza genea erabiltzen da kontrol gisa.
- Hiru birus-geneetatik gutxienez bi anplifikatzen dituzten eta RPP30 genea 35 ziklo baino gutxiago igarota anplifikatzen duten laginak positibotzat jotzen dira.
- Hiru birus-geneetatik bat ere anplifikatzen ez dituzten (Ct>40) eta RPP30 genea 35 ziklo baino gutxiago igarota anplifikatzen duten laginak negatibotzat jotzen dira.
- Negatibotzat edo positibotzat hartzeko irizpideak betetzen ez dituzten laginak zehaztugabetzat jotzen dira. Halakoetan, gomendatzen da beste lagin bat hartzea egun batzuk igaro eta gero.
- RPP30 genea 35 ziklo baino gehiago igarota anplifikatzen bada (edo anplifikatzen ez bada; Ct>40), lagina BERRIRO hartu behar da; izan ere, jotzen da ez duela analizatzeko besteko kalitaterik.
